Nous voici à la quatrième partie de l’IA dans la Santé

Le projet de recherche européen HosmartAI a pour objectif de doter l’Europe d’un système de santé fort, efficient, durable et résilient, bénéficiant des capacités d’IA des acteurs européens de la technologie pour générer un impact. Dans ce but, une plateforme commune d’intégration ouverte avec les outils nécessaires pour faciliter et mesurer les bénéfices de l’intégration des technologies numériques (robotique et IA) pour les professionnels de santé, les patients, les gestionnaires de systèmes d’information et les administrations des organisations de santé sera créée.
La mission du projet HosmartAI compte 4 objectifs principaux, divisés en trois catégories principales :
La plateforme sera validée par huit projets pilotes à grande échelle qui mettront en œuvre et évalueront les améliorations apportées au diagnostic médical, aux interventions chirurgicales, à la prévention et au traitement des maladies, ainsi qu’à la réadaptation et au soutien des soins de longue durée dans divers hôpitaux et établissements de soins.
Ces huit applications pilotes alimentées par l’IA, actuellement testées, impliquent 3 000 patients, 300 personnels de la santé et 600 intervenants de 5 hôpitaux en Grèce, en Espagne, en Italie, en Belgique et en Slovénie.
Ils pourraient à terme bénéficier au traitement du cancer, des troubles gastro-intestinaux et thoraciques, des maladies cardiovasculaires et neurologiques, des soins aux personnes âgées et de la réadaptation neuropsychologique, ainsi qu’au retard de croissance du fœtus et à la prématurité.
.
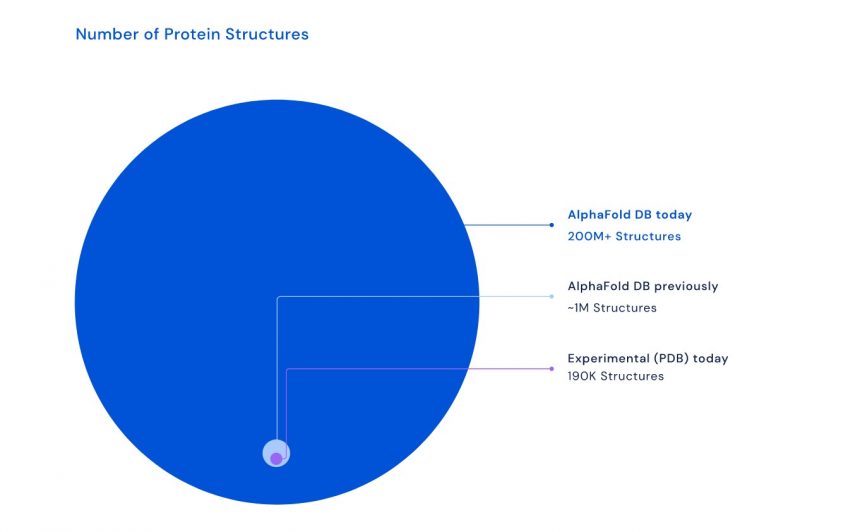
L’IA AlphaFold a pu prédire la structure moléculaire de millions de protéines, une avancée considérable pour la recherche en biologie.
DeepMind Technologies a dévoilé ce 28 juillet 2022 les progrès majeurs de son IA AlphaFold. Cette dernière a prédit la structure de presque toutes les protéines connues par la science. Le laboratoire a partagé la base de données d’AlphaFold qui contient plus de 214 millions de modèles en trois dimensions des protéines issues d’organismes vivants. Une avancée qui va permettre d’accélérer la recherche scientifique, notamment dans le secteur médical.
Le laboratoire DeepMind de Google n’en est pas à son premier exploit en matière d’intelligence artificielle. Les chercheurs qui y travaillent depuis sa création en 2010 et depuis le rachat par Google en 2014, ont conçu des IA capables de prédire les cancers du sein, les insuffisances rénales ou encore la météo. Le laboratoire s’est également fait connaître avec ses algorithmes qui rivalisent avec les plus grands joueurs du jeu de go ou de jeux vidéo.
AlphaFold, qui promettait de modéliser la structure des protéines, a vu le jour chez DeepMind en 2018. La première base de données d’AlphaFold a été lancée en juillet 2021 et contenait déjà 1 million de modèles de protéines, un nombre qui paraît bien dérisoire en comparaison des 214 millions de protéines que l’IA a pu prédire et modéliser seulement une année plus tard.
Il s’agit d’une avancée significative pour les chercheurs en biologie. Cette base de données qui contient des protéines du corps humain, ainsi que des protéines animales, végétales ou encore issues des bactéries, devrait par exemple accélérer la découverte de nouveaux médicaments et de vaccins.
Des chercheurs de l’université d’Oxford ont déjà utilisé AlphaFold pour mener leurs recherches sur la conception d’un vaccin contre la malaria, maladie qui fait des centaines de milliers de morts chaque année. Les scientifiques ont pu utiliser le modèle de structure de Pfs48/45, protéine de la malaria, prédit par AlphaFold et le comparer à une version moins précise issue de leurs propres expériences. Les deux modèles s’alignaient presque parfaitement pour créer une image nette de la molécule. Ils ont ainsi pu déterminer comment elle fonctionne et comment produire des antibiotiques avec celle-ci.
« II s’agit d’une réussite incroyable, à la fois pour la science et pour l’intelligence artificielle, démontrant son rôle en tant qu’outil au service des découvertes scientifiques et à une échelle jamais atteinte auparavant », a expliqué Shrikanth Narayanan, professeur à l’université de Californie du Sud, au Wall Street Journal. Maintenant que la base de données d’AlphaFold s’est étoffée, elle pourrait mener à de nombreuses découvertes médicales dans le futur.
Cet article est un résumé de : DeepMind : une IA a modélisé la quasi-totalité des protéines connues
Publié sur Siècle Digital par Maxime Mohr le 29 juillet 2022
Autre article sur ce sujet : L’IA AlphaFold prédit la structure de presque toutes les protéines connues
Publié sur Le Journal du Geek par Antoine Gautherie le 29 juillet 2022

La « medtech » SONIO vient d’obtenir une autorisation de l’agence américaine du médicament pour un logiciel qui potentialise l’efficacité de la prise d’images échographiques.
Sonio propose un logiciel d’aide au diagnostic Cloud, en temps réel et s’appuyant sur de multiples données.
Créé par une équipe d’experts scientifiques et médicaux, Sonio s’appuie sur une recherche et un développement continu, pour incorporer régulièrement de nouvelles briques technologiques à son logiciel.
Sonio Diagnostics accompagne les experts en échographie fœtale et diagnostic prénatal. Améliorez le diagnostic des anomalies fœtales et syndromes pathologiques associés à l’aide de l’Intelligence Artificielle.
Sonio Pro accompagne tous les spécialistes de l’échographie fœtale : sages-femmes, gynécologues-obstétriciens et échographistes. Sécurisez les examens de routine et améliorez le dépistage prénatal grâce à une checklist intelligente.
Sonio Share accompagne tous les spécialistes de l’échographie fœtale : sages-femmes, gynécologues-obstétriciens et échographistes. En 1 clic, partagez ou imprimez les échographies foetales pour votre patiente.
« Vous voyez cette pile de bouquins ? Il y a plus de 400 syndromes prénataux référencés là-dedans. Le problème, c’est que tout cela ne rentre pas dans un cerveau de médecin, même le meilleur ! » lance le Pr Yves Ville, chef de service de la maternité de l’hôpital Necker-Enfants Malades, à Paris, en désignant une impressionnante colonne de livres de médecine obstétrique qui court du sol au plafond de son bureau. « Un spécialiste en néonatalogie emmagasine, au mieux, une cinquantaine de pathologies parmi les plus fréquentes. C’est un cerveau augmenté qu’il lui faut, pour gérer cette masse d’informations. »
Sonio, système d’intelligence artificielle spécialisé dans la santé prénatale de l’enfant et de la femme enceinte, qu’il a cofondé, est une sorte d’extension de son cerveau et de celui des autres praticiens. Né en 2020 de la collaboration entre l’hôpital Necker et l’école polytechnique, Sonio apporte une solution de contrôle aux gynécologues-obstétriciens, médecins généralistes et échographistes pendant le suivi de grossesse et leur permet de mieux détecter in utero les potentielles anomalies.
Lire également : La medtech française Sonio met l’IA au service de la santé prénatale Publié sur Siècle Digital par Mathilde Rochefort le 1er février 2024
Le nouvel environnement logiciel de travail développé par Microsoft EvoDiff fonctionne grâce à la puissance de l’Intelligence Artificielle ; il sera utilisé pour améliorer le processus de protein design.
On le sait depuis quelque temps maintenant, l’IA générative est en train de bouleverser l’économie mondiale, avec de nouvelles méthodes de travail dans divers domaines. Plusieurs solutions logicielles ont déjà été développées pour améliorer la conception de protéines en laboratoire : AlphaFold de Google, FoldX. Là où EvoDiff innove, c’est qu’elle peut générer des protéines à partir simplement d’une séquence donnée, ce qui simplifie grandement le processus. Traditionnellement, il fallait des informations précises sur la structure tridimensionnelle cible de la protéine souhaitée afin de prédire correctement la manière avec laquelle la séquence d’acides aminés se replierait pour être fonctionnelle.
En éliminant cette étape, le gain de temps et d’argent est énorme. Le chercheur principal de Microsoft, Kevin Yang, imagine déjà les applications potentielles d’EvoDiff dans différents domaines : création de nouvelles enzymes, usages industriels ou thérapeutiques…
EvoDiff doit principalement son efficacité au très grand nombre de paramètres dans lesquels il est capable de piocher : 640 millions. Tous sont formés grâce à des bases de données issues de différentes classes fonctionnelles ou types de protéines existantes. Le système fonctionne grâce à un modèle de diffusion. Grâce à cela, il déduit et crée efficacement des séquences de protéines en éliminant le « bruit », c’est-à-dire toute information inutile ou non pertinente qui viendrait perturber le processus de conception.
Le système développé par Microsoft semble très prometteur, mais son efficacité n’a pas encore été clairement évaluée par des acteurs scientifiques extérieurs. Si celle-ci est un jour prouvée, EvoDiff marquera alors un grand pas dans les progrès de la bio-informatique.
Article en anglais sur EvoDiff : Microsoft open sources EvoDiff, a novel protein-generating AI
Publié sur TechCrunch par Kyle Wiggers le 14 septembre 2023
EvoDiff est en open source : microsoft / evodiff
Une banale fracture et un algorithme ont transformé l’été 2019 de Frances Walter, une octogénaire américaine, en véritable cauchemar. Son histoire est le point de départ d’une vaste enquête qui montre comment des assureurs utilisent, aux États-Unis, un algorithme, pour décider de la durée de la prise en charge de leurs assurés.
Quand l’Intelligence artificielle et la santé ne font pas bon ménage… C’est à peine le début de l’été dans le Midwest des États-Unis lorsque Frances Walter, 85 ans, se fracture l’épaule gauche. L’octogénaire, qui vit seule, appelle les urgences. Elle est envoyée dans un hôpital du Wisconsin, et n’avance aucun frais pour y être soignée : elle a la chance d’être bénéficiaire de la « Medicare Advantage », un système d’assurance sociale et de complémentaire gérée par un assureur privé aux États-Unis. Sa convalescence sera longue, trop longue. Voilà en tout cas ce que pense l’algorithme utilisé par son assureur, qui estime qu’au 16e jour de séjour postopératoire, la patiente peut rejoindre son domicile. Le paiement des soins est interrompu au 17e jour. Problème : le corps médical préconise plutôt un séjour de trois semaines minimum, notamment parce que la vieille dame est allergique aux analgésiques et qu’elle se remet moins vite que prévu. Et ce n’est pas non plus ce que pense l’octogénaire, qui au 17e jour ressent toujours d’intenses douleurs. D’ailleurs, elle est toujours incapable de s’habiller seule ou de pousser un déambulateur.
La patiente est alors contrainte de puiser dans ses économies – la somme monte à des milliers de dollars aux États-Unis – pour être soignée jusqu’à un complet rétablissement. Ce montant lui sera, heureusement, remboursé, mais seulement… un an plus tard, après d’innombrables procédures et la décision d’un juge fédéral. Vous pensez que cette histoire est irréelle ? Elle a pourtant bien eu lieu pendant l’été 2019.
Et elle est loin d’être un cas isolé, rapporte le site d’informations Stat News qui publie, le 13 mars, une vaste enquête sur ce sujet.
Aux États-Unis, il n’est pas rare que les compagnies d’assurance rejettent des demandes de remboursement de frais médicaux. Mais ce serait désormais l’intelligence artificielle (IA) qui serait à l’origine de ces refus, rapporte le média américain, listant spécifiquement des cas de bénéficiaires de la « Medicare Advantage » qui couvre plus de 31 millions de personnes dans le pays. Certains assureurs privés, qui gèrent cette assurance sociale destinée aux plus de 65 ans aux revenus faibles ou modérés, utiliseraient un algorithme prédictif. Ce système d’IA déterminerait à quel moment l’assureur peut interrompre le paiement des séjours postopératoires des patients, au grand dam des médecins et des premiers concernés. En cas de désaccord, les malades doivent avancer les frais, faire appel et attendre des mois, voire des années, pour espérer être remboursés. Certains, qui ne disposent pas de telles sommes, se passent de soins, à l’image de ce patient de Caroline du Nord.
Le retraité n’avait pas pu être transféré dans un centre de soins postopératoires après un AVC. Brian Moore, son médecin, lui avait rendu visite à son domicile, faute d’avoir réussi à convaincre son assureur de lui payer ce séjour. « Il était assis, il essayait de se nourrir. Il disait : “Je ne pensais pas, lorsque j’ai opté pour la Medicare Advantage, que je ne pourrais pas obtenir les soins dont j’ai besoin”, raconte-t-il à nos confrères. Il bavait et pleurait. »
Tout aurait réellement commencé en 2020, quand UnitedHealth, le plus grand assureur Medicare Advantage du pays, rachète NaviHealth, une société de gestion de données médicales dont le produit phare est un algorithme appelé nH Predict. En se basant sur l’âge, les diagnostics des médecins et les informations médicales d’un assuré, et en les comparant avec des profils similaires dans une base de données de 6 millions de patients, l’algorithme rend un rapport dans lequel sont décrits les besoins médicaux, la durée estimée du séjour hospitalier et de la date cible de sortie d’un patient.
Cet algorithme « n’est pas utilisé pour déterminer la couverture médicale », a pourtant insisté UnitedHeatlh, qui n’a pas souhaité répondre aux questions de nos confrères, mais qui a publié un communiqué. Cet « outil » est utilisé comme « un guide », a ajouté la société. Ce n’est pas l’avis des soignants interrogés par Stat News. Pour ces derniers, les refus de prises en charge auraient commencé à se multiplier à partir du moment où UnitedHealth a été aux manettes de nH Predict. L’algorithme, qui était présenté comme un moyen d’éviter les soins inutiles et la surfacturation, irait encore plus loin que les années précédentes. Et désormais, les refus de prise en charge de UnitedHealthcare et de NaviHealth seraient devenus la norme. Y compris lorsque les patients sont manifestement plus malades que ce que prévoit l’algorithme, rapporte le directeur d’un établissement de soins post-intensifs à nos confrères. Pour preuve, le nombre de recours des patients entre 2020 et 2022, pour contester les refus de demandes de prises en charge, a augmenté de 58 %, selon une base de données fédérale. Et dans la majorité des cas, le juge tranche en faveur du patient, et ordonne un remboursement.
Autre problème soulevé par cette enquête : l’opacité de l’algorithme. À chaque fois qu’un médecin ou directeur d’hôpital demande des explications sur la durée excessivement courte d’une prise en charge par l’assureur, la réponse est toujours la même : la boîte noire de l’IA est devenue une excuse générale pour justifier les refus de paiement, expliquent nos confrères. Même topo du côté des patients. La fille de Dolores Millam, 89 ans, qui s’est vu refuser la prise en charge d’un séjour en maison de santé médicalisée après une fracture, témoigne : « Lorsque vous essayez d’appeler et de raisonner quelqu’un, lorsque vous voulez obtenir des explications, vous finissez par parler à un service basé aux Philippines. Pour eux, il s’agit simplement d’une question de procédure qui n’a rien à voir avec les soins ». UnitedHealthcare s’abriterait derrière les lois de protection de la vie privée pour refuser toute discussion avec les familles des patients.
Car pour l’instant, la loi n’impose pas la moindre transparence aux assureurs qui utilisent un algorithme. Ils ne sont ni contraints de rendre publiques les données des études d’efficacité, ni obligés de respecter un code de conduite éthique – du moins, pas encore. Le législateur américain a en effet publié, en 2022, un projet de charte des droits de l’IA qui vise à protéger les citoyens américains d’usages dangereux ou discriminatoires d’IA. Mais ce texte n’est pour l’instant pas contraignant. En Europe aussi, le futur règlement sur l’Intelligence artificielle, actuellement en discussion au Parlement européen, prévoit des obligations très strictes en cas d’utilisation de l’IA dans la santé.
Pourtant, un contrôle de système d’intelligence artificielle dans ce secteur existe déjà, mais à la marge. Les modèles d’IA utilisés par le corps médical, qui permettent par exemple de diagnostiquer des cancers, doivent être, avant toute utilisation, approuvés par la FDA – l’équivalent de l’Agence de sécurité du médicament aux États-Unis. Alors pourquoi pas cet algorithme, qui a des conséquences plus qu’importantes sur la santé de milliers d’Américains ?
Article source : Quand l’IA est utilisée pour écourter la durée de séjours hospitaliers
Publié sur 01Net par Stéphanie Bascou le 16 mars 2023 à partir de : Stat News
Mise en garde l’OMS : un déploiement rapide des programmes de santé numérique pourrait porter préjudice aux patients
L’intelligence artificielle peut transformer les traitements médicaux, mais sans une compréhension de son fonctionnement, un déploiement rapide des programmes de santé numérique pourrait porter préjudice aux patients, a mis en garde jeudi 19 octobre l’OMS.
«Avec la disponibilité accrue des données sur les soins de santé, et les progrès rapides des techniques analytiques – capacités d’apprentissage automatique des ordinateurs, basées sur la logique ou les statistiques – l’IA peut transformer le secteur de la santé», a indiqué l’Organisation mondiale de la santé dans ce document consacré à l’utilisation de l’IA, destiné à donner des orientations aux pouvoirs publics.
Selon l’OMS, l’IA peut améliorer les performances pour ce qui concerne les essais cliniques, le diagnostic, la mise au point d’un protocole de traitement, et intervenir en complément des connaissances et compétences médicales. Par exemple l’IA s’avère utile en l’absence de spécialistes, dans le domaine de la radiologie pour l’interprétation de l’imagerie médicale et des images rétiniennes. Cependant, l’IA est déployée rapidement, parfois sans compréhension adéquate de la manière dont fonctionnent ces technologies, «qui peuvent soit bénéficier soit être préjudiciables aux utilisateurs», qu’il s’agisse des patients ou des professionnels, a averti l’OMS.
Les systèmes d’intelligence artificielle appliqués à la santé permettent l’accès aux données personnelles, c’est pourquoi un cadre légal solide est nécessaire pour sauvegarder la vie privée, a souligné l’OMS. «L’intelligence artificielle est très prometteuse pour la santé, mais présente aussi de sérieux défis, dont la collecte non éthique de données, des menaces pour la cyber-sécurité, et l’amplification des préjugés et de la désinformation», a déclaré le directeur général de l’OMS. «Ce nouveau guide aidera les pays à réguler d’une manière efficace l’IA, à exploiter son potentiel, qu’il s’agisse de traiter les cancers ou de dépister la tuberculose, tout en minimisant les risques», a-t-il ajouté.
Selon l’OMS, les systèmes d’intelligence artificielle dépendent des données d’entraînement qu’ils utilisent pour leur apprentissage, c’est pourquoi une meilleure règlementation peut contribuer à gérer les risques et le danger de voir les préjugés présents dans les données existantes amplifiés par l’IA. «Par exemple, il peut être difficile pour les modèles de l’IA de représenter d’une manière correcte la diversité des populations, ce qui conduit à des préjugés, des inexactitudes et même des échecs», a précisé l’OMS.
«Pour contribuer à réduire ces risques, des règlementations peuvent être utilisées pour s’assurer que les attributs, comme le genre, la race et l’origine ethnique des personnes figurant dans les données d’entraînement soient signalés et que l’ensemble des données soit intentionnellement conçu pour être représentatif», ajoute l’OMS. L’organisation recommande dans son guide une validation externe des données, l’évaluation des systèmes afin d’éviter les erreurs et les préjugés, le consentement pour le recueil des données privées, et la collaboration entre régulateurs, patients, gouvernements et professionnels de santé.